U-Net: Convolutional Networks for Biomedical Image Segmentation
2021/7/13
来源:MICCAI15
resource:github上备份的包括ipad标注的pdf版本。
作者是University of Freiburg的Olaf Ronneberger, Philipp Fischer和Thomas Brox,这篇文章属于开宗立派的鼻祖文章了,单篇引用2.8w+!
Summary:文中提出了一种新颖的网络结构,因为特征提取与恢复部分呈镜像对称而命名为U-Net,这篇文章(的网络结构)从医学图像分割一路杀穿了图像分割、图像去噪等领域,成为魔改结构的经典(?)。
rating:5.0/5.0(不要不识抬举)
comprehension:3.5/5.0
文章的贡献有:
- 提出了一种新颖的全卷积网络结构U-Net,只需要非常少的数据配合数据增强就能取得非常理想的分割性能,后来在医学图像分割等领域取得了辉煌战果;
1 Introduction
- 图像分割输出的通道数和分类数目一致,本质上是个per-pixel生成label的过程。
网络结构如下所示:
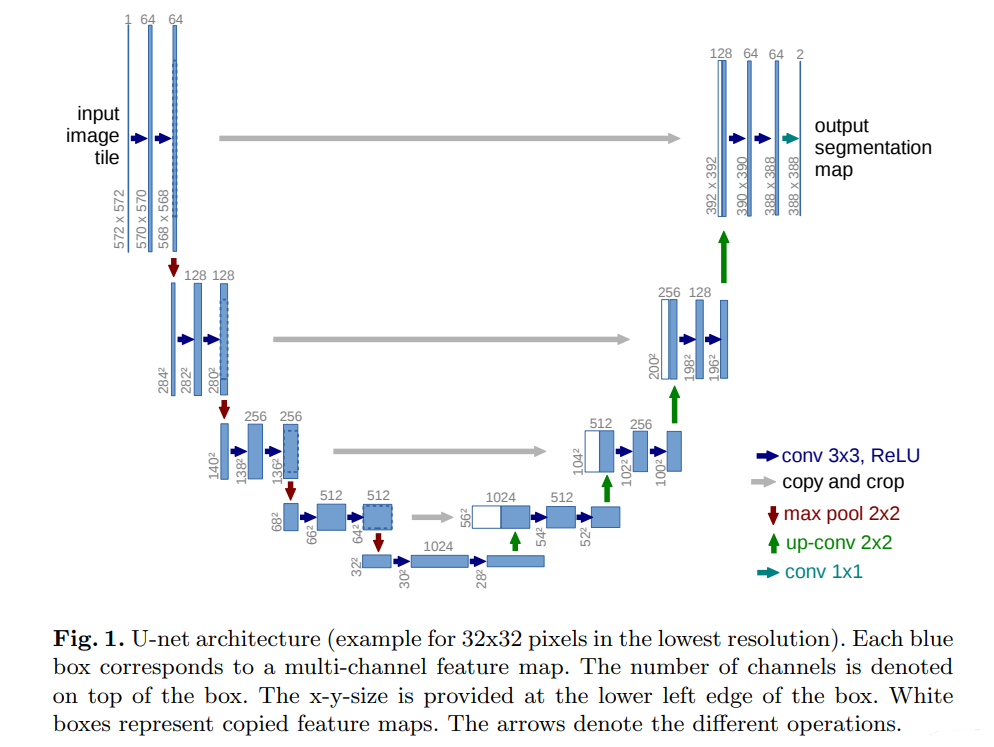
U-Net由两块组成(这两块分别可以看成Encoder和Decoder),分别取名叫contracting path和 symmetric expanding path,前者提取语义信息,后者用来定位。卷积采用有效卷积,因此每次卷积后feature map的尺寸都会缩小,需要精心设计feature map的尺寸(后续的工作有觉得比较麻烦,所以直接换成Same Conv了),在两条path之间连有skip connection,将前面的信息保留到后面(这里有个尺寸的对齐)。采用的降采样方法是max pooling,采用的升采样方法是up conv。网络中没有全连接层,是个全卷积神经网络。
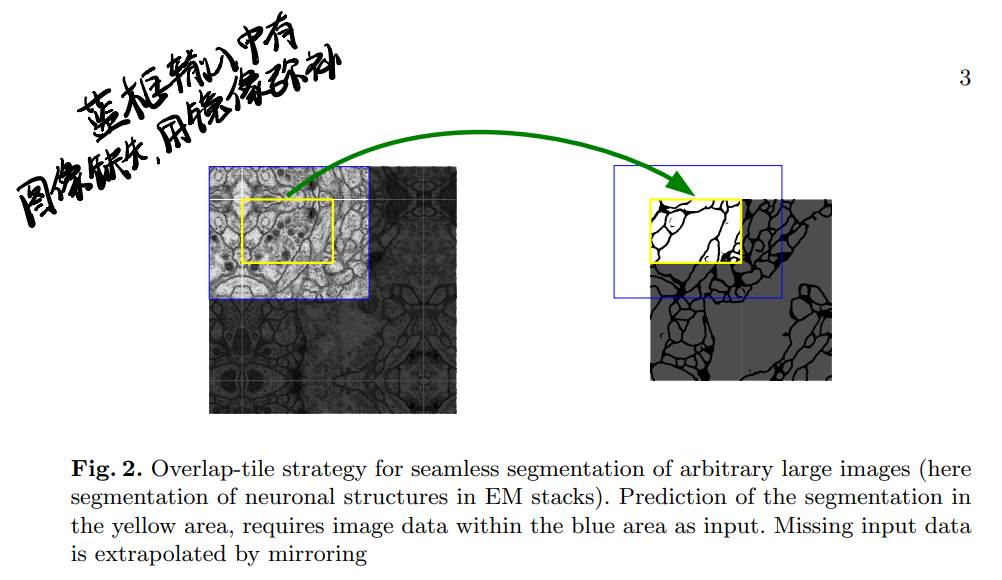
网络有个tile-strategy,因为有效卷积生成的feature map只占输入图像的一小部分,所以对大图像进行分割时就要在有效部分外留足额外图像,这对于中间的分块来说比较容易满足,但是边缘分块则需要用镜像来填补(不补0,充分利用图像信息),但是这个策略是因为有效卷积产生的,替换掉有效卷积是不是就不用这么麻烦了?而且去噪的话少一点边信息应该问题不大?
此外,为了解决数据量不够的问题,作者使用elastic deformations作为数据增强的方法,这对于细胞来说还是比较合适的。
为了分辨出细胞的边界,作者提出对细胞边界的像素乘以一个较大权值,这一部分不是很重要(有一些细节也没懂),用截图放出:


最后是些训练设置等等细枝末节的东西。